基于微小RNA的心血管疾病的诊断与治疗
1993年,研究者开始发现小RNAs的功能和重要性,而且因它们在控制人类基因表达的能力(1),称之为“微小RNAs”(microRNAs,miRNAs)(2)。之后,miRNAs便吸引了科学家们的注意,究其原因在于在各种疾病的诊断与预后中,它是一个比较明显的生物标志物(3-5)。而且,miRNAs在疾病模型中也被当作是一种潜在的基因特异性治疗靶点(6)。
在心血管疾病中,miRNAs被认为是疾病特异性生物标志物,而且在某种疾病中会发生定量改变,例如心肌梗死(myocardial infarction,MI),动脉粥样硬化,冠状动脉疾病(coronary artery disease,CAD),心力衰竭,心房颤动,心肌肥厚及纤维化(7-10)。
在本文中,我们探讨心血管miRNAs(尤其是在CAD及MI中)的价值,并对miRNAs用作心血管疾病的诊断与预后的生物标志物以及治疗药物开发进行概述。
miRNA-概述它们是什么
miRNAs是一类含量丰富的非编码RNAs分子,由19-25个核苷酸组成(11,12)。miRNAs在转录后水平阻遏翻译或者降解信使RNA(messenger RNAs,mRNAs),负向调节基因表达,从而影响各种各样的细胞进程(13-15)。2588个成年人类miRNAs被列于“miRBase”中(16)。计算预测靶mRNAs发现超过60%的哺乳动物的蛋白编码基因是miRNAs保守靶点(17),而每个单独的miRNAs具有数百个不同基因的靶位点(18)。miRNAs研究揭示其组织特异性表达(19),也显示病理中浓度依赖性对器官及组织的影响(20)。
miRNAs在心血管疾病,尤其是CAD中作为一种有前景的新生物标志物的发现引发广泛的关注。miRNA定量研究表明miRNAs能够在组织及细胞特异性表达(19),体外研究结果表明许多miRNAs在心血管疾病的不同领域发生特异性地上调和下调(21),而miRNA调控路径的多态性—谓之miRSNPs—被证实与不同疾病相关(22-24)。因此,miRNAs符合成为一种理想的生物标志物的几个标准。循环的稳定性,组织与病理特异性调控以及高度敏感性及特异性表明miRNAs作为心血管疾病的生物标志物的适用性甚至超过了基于蛋白质的生物标志物(25,26)。miRNA研究已经为心血管事件提供一种新的可能以生物标志物为基础的危险分层方式,而且在大规模的前瞻性临床研究中收集了有前途的数据(27)。
CAD的生物标志物
具有发生MI高风险的稳定性CAD的诊断是通过有创冠状动脉造影来证实的。研究证实CAD患者较健康者特定的miRNAs水平发生显著性改变,提示miRNAs可能成为CAD的诊断性生物标志物(7,21,28)。
新开发的高敏性心脏肌钙蛋白(high sensitive cardiac troponin,hs-cTn)的高敏感度(29,30) 引起非冠状动脉心脏病的假阳性增加(31)。而且,急性心肌梗死(acute myocardial infarction,AMI)的早期诊断仍受限于肌钙蛋白的延迟释放。因而,需要一种高敏感性和高特异性生物标志物,用以早期发现AMI,从而进一步降低AMI的死亡率(32)。而且,也缺乏一种生物标志物能够鉴别钙蛋白阴性的不稳定型心绞痛(unstable angina pectoris,UAP)和非冠心病性胸痛(non-coronary chest pain,NCCP)。在心血管疾病的急性背景下,大量研究证实miRNAs能够成为MI的生物标志物(33-35)。
CAD和MI的miRNAs
通过调控涉及内皮细胞(endothelial cell,EC)功能的信号通路,miRNAs及其关键酶Dicer在ECs的功能中发挥重要作用(36)。Dicer敲除诱导ECs中高表达miRNAs(let-7,mir-21,mir-126,mir-221,及mir-222)调节障碍,导致EC增殖受损(37) ,并使炎症趋化因子和细胞因子(如IL-8和IL-1β)以及趋化因子配体1和3表达减少(38)。这些观察结果强调了miRNAs参与到动脉粥样硬化及CAD的发病过程中。
在斑块形成的过程中,不同的miRNAs在斑块进展的不同阶段出现(39),而且miRNAs调节障碍在动脉粥样硬化斑块的去稳定性和破裂中起着至关重要的作用(40)。
血管的miRNA
体外研究表明几个miRNAs特异性影响血管新生和单层内皮细胞的完整性(37)。尤其是miR-27b和miR-130a能够促进血管新生,而miR-221和miR-222却抑制血管新生,从而它们共同参与动脉粥样硬化及CAD的发病过程并被检测到(41,42)。事实上,体外研究证实miR-221及miR-222能够调节其他miRNAs,其中包括miR-223(43)。动脉粥样斑块形成涉及炎症反应,而且在很大程度上受到体内胆固醇稳态的影响(44)。在小鼠模型中,Vickers等人证实miR-223能够调节胆固醇生物合成、吸收及排出过程(45)。这表明它是动脉粥样硬化潜在的调解者。而且,他们还发现miR-223在动脉粥样硬化患者的血小板中表达水平升高。同时,与血管生成相关的miRNA-106b,-25,-92a及-21也在动脉粥样硬化斑块的颗粒中显著增加(46)。尤其是miR-92a在小鼠模型及人类脐静脉来源的内皮细胞中被证实是血管生成的重要的调控因子(47)。有趣的是,不同的研究发现miR-92a具有促进动脉粥样硬化作用,以及体内外实验均表明内皮细胞中过miR-92a的过度表达能够阻止血管新生(47,48)。
miR-143和-145是血管平滑肌(vascular smooth muscle cells,VSMCs)高度表达的miRNAs,能够调控VSMCs的分化和功能(49)。在体外模型中,它们在血管应激下表达降低,表明它们在血管疾病的血管平滑肌细胞表型调节中发挥作用(50)。研究表明miR-143和-145在血管损伤后的血管内膜形成中发挥至关重要作用,这证明了它们在动脉粥样硬化疾病中的重要性(51)。
除了miR-142及-145之外,miR-126被认为是血流敏感性miRNA,能够调节内皮细胞的基因表达,同时参与正如CAD发生时血流改变所致的血管内皮细胞的功能障碍和动脉粥样硬化过程(52)。
心肌的miRNAs
miR-1是心脏和骨骼肌中含量最丰富的特异性miRNA,对于心脏发育起着调节分化及增殖的作用(53)以及对成人心脏心肌细胞生长起到调节作用(54)。同时,miR-1被证实能够促进缺血所致的心肌细胞凋亡(55)。在大鼠模型中,miR-1在冠脉闭塞12小时后心肌中的表达显著性增加(56)。而且,因MI死亡的患者miR-1表达在梗死心肌组织和健康成人心肌中显著性增加(57)。
miR-133是通过miR-1相同染色体位点转录而来,能够增加成肌细胞增殖从而参与心肌细胞增殖(58)。然而,有趣的是miR-1通过氧化应激能够促细胞凋亡,miR-133被发现能够抑制caspase-9表达从而起到抗凋亡作用(55)。
miR-21在心肌细胞缺血后立刻出现表达上调,直到心肌细胞死亡后表达下降(59)。然而,miR-21在成纤维细胞中表达增加促进细胞增殖,表明miR-21参与重塑过程(59)。
小鼠动脉模型研究证实miR-208仅在心肌细胞表达,而且在MI的心肌细胞死亡时释放(60)。
miR-29家族成员在小鼠和人类临近MI区域的心肌组织的表达能够降低(61)。而且,作者还证实miR-29在心肌纤维化中起到的调节作用,有望成为组织纤维化的治疗靶点。
miR-126在诱导所致的大鼠心肌梗死模型中的非梗死心肌组织区域表达增加(62)。同时,另一个研究证实miR-126敲除小鼠较野生型小鼠在接受永久性冠脉闭塞时幸存率明显降低(63)。作者认为这与miR-126敲出小鼠的血管新生能力缺陷。miR-126被认为主要是通过促进血管新生参与心肌损伤后的修复过程(59)。
Tang等研究缺血再灌注对大鼠心脏miRNA的影响(64)。结果发现miR-1、miR-126和miR-208表达增加,而miR-21、miR-133和miR-195表达降低。Botjancic及同事对50名死于MI的患者及八名以往健康的创伤受害者的心肌组织样本进行分析(65)。结果发现与健康对照组相比较,梗死组织中miR-208水平上调而miR-1和miR-133a/b下调。
体液的miRNAs及其作为生物标志物的应用
细胞内的miRNA已被证实在病理条件下对于新陈代谢能够起到调节和介质的作用(66)。生理条件下,细胞间miRNAs提供基因调控及表型调控(67)。既然miRNAs在许多体液中被稳定地检测到,如血清、血浆、唾液、眼泪及尿中(25,26),因此miRNAs作为细胞间交流的细胞外信号以及作为心血管疾病潜在循环生物标志物的重要性在过去的几年已经被证实(3,68)。
循环miRNAs存在于胞外体、微泡、凋亡小体—所谓的微粒中(69,70),也存在于RNA结合蛋白(71)或HDL脂蛋白复合物中(72)。这些结构条件被认为是造成循环血中miRNAs特别稳定的原因(25,73),同时证实了能够在体液中检测到miRNAs的一个先决条件。miRNAs释放入细胞室,再进入体液,特别是进入血液中,这一过程对无创性检测那些miRNAs以及用作为疾病生物标志物提供可能性。许多生物标志物领域的研究都是基于循环miRNAs的量化。
急性MI诊断中的循环miRNAs
后来,为了证明miR-208在大鼠心脏中特异性表达,Jiet等人采用异丙肾上腺素诱导的大鼠MI模型发现MI后的miR-208血浆水平显著增高(74)。作者得出一种可与心肌肌钙蛋白时间因素检测限制相似的结论,那就是miR-208对于MI的检测可能是一种有用的循环生物标志物。这些也来自不同研究小组的研究结果,并发现冠状动脉闭塞的大鼠血浆中miR-208水平增高(74)。对于407名疑似ACS的患者血浆中,miR-208b及miR-499-5p被检测到并能够成功的鉴别诊断MI(74)。与肌钙蛋白T相比,AUC的识别能力是相当低的(cTnT=0.95,miR-208b=0.82,miR-499-5p=0.79)。
这些发现立足于Adachi等对于不同人类组织利用miRNA数组分析去确定心脏特异性miRNAs,并发现miR-499几乎特异性表达于心脏(77),表明miR-499对于MI可能为一种额外的有前景的生物标志物。类似的结果也在92名非ST段抬高性心肌梗死(non ST segment elevation myocardial infarction,NSTEMI)的老年患者中报道。作者发现与90名对照组及81名急性心衰而无MI的患者相比,miR-499-5p水平增长超过80倍(78)。miR-499-5p区分NSTEMI的能力可与cTnT相媲美。更重要的是,miR-499-5p较心肌肌钙蛋白更能区分急性心衰和NSTEMI。这一观察表明,miRNAs可能是一种潜在有用的生物标志物,能够降低非冠状动脉粥样硬化心脏病中心肌肌钙蛋白的(尤其是hs-cTn)假阳性率。
在诱导的大鼠心肌梗死模型的血清中miR-1被发现增长200倍,峰值为6小时,其水平与梗死面积和CKMB水平紧密相连(79)。而来自93名AMI患者及66名健康者的一项研究表明与对照组相比较,AMI患者的血浆样本中miR-1水平显著增加(80)。这些数据也被Wang等人对33名AMI患者,30名健康者,16名稳定型冠心病及伴有其他心血管疾病的17名患者进行的研究所证实(75)。同样地,相似的血浆miRNA水平也在444名ACS的患者中被检测,而且与UAP相比较,MI中miR-1,miR-133a,miR-208b的表达水平显著增加(81)。有趣的是,miR-1,miR-133a/b,miR-208b均与高敏肌钙蛋白T水平独立相关。Li及他的同事还发现与32名健康着的对照组相比较67名AMI患者血浆中miR-1,miR-208b及miR-499水平显著增高(82)。出院患者miRNAs的表达已降至基线血浆水平之下。而且,ROC曲线分析显示这四种miRNAs在预测MI方面不劣于肌钙蛋白T。
这些数据表明一些miRNA在缺血和MI时调节失衡,而且对于MI的诊断似乎是适合的生物标志物。表1给出了MI中循环miRNAs失调的概述。
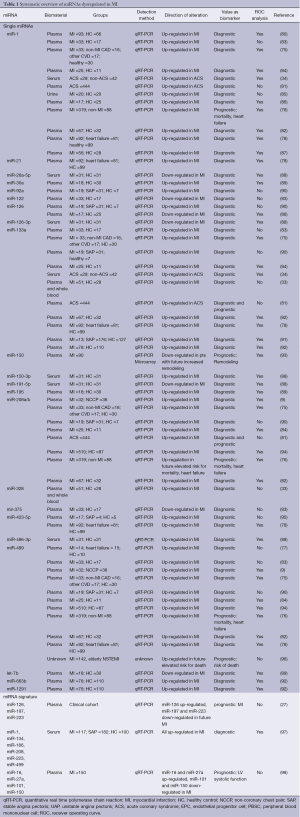
Full table
最近,Cheng等人进行了一项荟萃分析得出结论是miRNAs适合成为诊断MI的生物标志物(99)。十五项研究报告报道了有关MI中敏感性、特异性及AUC分析,四项研究没有报告这些发现。他们利用整体miRNA分析发现一种敏感性为0.78,特异性为0.82,AUC面积为0.91的miRNA来鉴别MI。四项有关miRNAs大型报告的亚组分析(miR-1,miR-133a,miR-208b及miR-499)证实了这些结果,但是miR-1表现出特别低的敏感性及特异性。而且,作者还发现miRNAs与MI其他诊断性生物标志物例如cTnT,hs-cTnT,CK,CK-MB之间的高度相关性。
与血液样本miRNA定量来诊断MI相比较,其他体液研究的相关报道是罕见的。有研究发现与20名健康者的对照组相比较,20名STEMI患者尿中的miR-1水平显著提高(85)。而且值得注意的是,尿中miR-1水平与血清cTnI和CK-MB水平密切相关。
稳定性CAD诊断中的循环miRNAs
首个于临床背景下进行人类循环miRNAs在CAD中的研究是Hoeckstra等人于2010年进行的(100)。在50名有创确诊的CAD患者中,157个检测的miRNA中的129个在外周血单核细胞(peripheral blood mononuclear cells,PBMCs)中表达。与健康对照组相比较,miR-135a及-147被发现有显著的升高。他们的研究结果表明血液循环中miRNAs可作为生物标志物用于检测CAD。另一个早期病例对照研究证实与63名健康对照组相比较,163名CAD患者的血液基因表达谱中miR-140-3p,-182水平降低(101)。
Fichtlschere等进行一个miRNA的筛选研究发现与17名健康对照人员相比确诊CAD的36名患者的血清和血浆中miRNAs发生了改变(7)。有趣的是,无论是内皮细胞表达的(miR-126,miR-92a,miR-17)或者平滑肌细胞(miR-145)及炎症细胞衍生物(miR-155)中miRNAs都显著的减少。同时,心肌细胞富集的循环miR-133及miR-208在CAD患者中升高。如前所述,miR-126,miR-92a及miR-145在小鼠动脉粥样硬化及体外模型中被证实是有用的指标。
Weber等人对与15名健康对照相比较的10名CAD患者的整个血液样本进行miRNA水平分析(102)。他们发现内皮miR-126及miR-92a没有像Fichtlscherer等人报道的那样出现定量的变化。但就如Fichtlscherer等人所说的那样,检测到miR-145及miR-155有所改变。作者讲述的差异是血液样本的基本差异。Weber等人用的是全血,而Fichtlscherer等人分析的是血清及血浆样本。这些表明在不同的血样本来源中检测的miRNAs也有很大不同,同时考虑到将miRNAs作为潜在的诊断生物标志物时,这些差异变得十分重要。
CAD患者miR-155水平失调在与22名健康者对照相比较的65名CAD患者中被证实,并发现CAD患者中miR-155及miR-146a也显著性失衡(103)。miR-146a水平的再次升高也在最近的一项病例对照研究中被报道,该研究涉及295名CAD患者及283名健康对照者(104)。南非患者中pre-miR-146a变异的显著高水平与CAD相关(105)。最终,与健康者对照相比的40名CAD患者中利用血小板衍生的miRNAs来分析富含血小板的血浆(106)。有趣的是,并没有像之前描述的那样两组之间动脉粥样硬化失调的血小板相关的miRNAs存在定量改变。两个独立验证队列研究证实CAD患者中miR-340及miR-634水平是上调的。这些有关失衡的循环人类miRNAs的研究表明它可作为生物标志物去诊断CAD。表2中给出了更详细的信息。
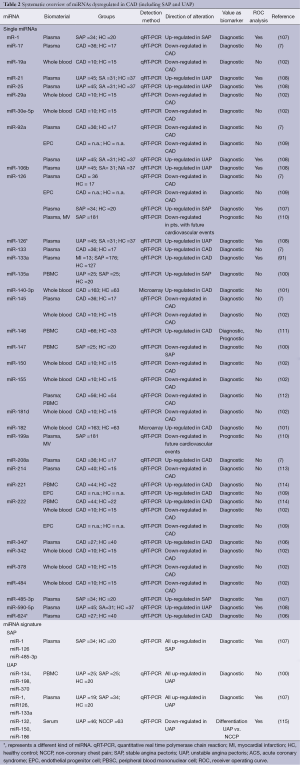
Full table
稳定型和不稳定型心绞痛诊断中的循环miRNAs
UAP的定义包括心肌肌钙蛋白的正常值。因此,不稳定型心绞痛的诊断是基于临床评估,但实验室指标仍然缺乏。然而,因为UAP病人MI的风险升高,所以UAP的诊断是至关重要的。Hoekstra等人对25名UAP及25名稳定型心绞痛(stable angina pectoris,SAP)的患者的miRNA水平进行比较(100)。结果发现三个miRNAs(miR-134,miR-198,miR-370)在UAP中过度表达,表明循环miRNAs在确定患者的MI风险方面可能是一种有效的生物标志物。这些研究符合有关微粒得到促炎性miRNA的描述并发现对10名稳定性CAD及UAP的患者血浆有鉴别能力(116)。
然而,D’Alessandra等人在与34名SAP患者比较的19名UAP患者中未能证实这些研究结果(107)。为了证明同样的问题,Zeller等人对于NCCP的患者和UAP患者进行了miRNA筛查与重复。第三步是作者评估与63名NCCP患者相对比的46名UAP患者中的miRNA水平(115)。结果表明miR-132,miR-150及miR-186的3-miRNA板对于UAP和NCCP具有很强的辨别能力。表2包括涉及SAP及UAP中miRNA的研究结果。这些将是有前景的结果,可以缩小鉴别诊断稳定性CAD和肌钙蛋白阴性的ACS患者基于生物标志物的差距。
miRNAs对CAD 和MI的预测和预后的价值
对于心血管疾病,尤其是CAD 和MI,miRNAs除具有诊断价值之外还可作为其预测生物标志物。
在444例ACS患者的单变量方差分析以及校正年龄和性别因素后的分析中,miR-133a和miR-208b的血浆水平与死亡风险有显著的相关性(81)。Hoekstra 等人检测出一个miRNA标志物能够辨别出不稳定型心绞痛及稳定型心绞痛,此标志物由三个miRNAs组成,分别是:miR-134,、-198、-370。这意味着miRNA有预测心血管未知风险时间的潜能并有可能成为临床疾病预后的一种手段 (100) 。miR-370在急性冠脉事件的危险分层中作为一个潜在的预后标志物是根据早期小鼠研究中类似的结果而得出的。在诱发的缺血事件中,miR-370表达水平上升(117)。在一项包括424例疑似MI研究中,升高的血清miR-208b、mir-499-5p水平均与30天内的死亡率或心脏衰竭的风险增加密切相关(76)。研究提供了不同水平的miRNA与心肌梗死结局关系的证据,然而校正TnT后,这种关系消失了。近日,一个筛选及验证miR-652的方法被发现了。miR-652水平与由于心衰心肌梗死后所致的住院率有显著相关性,同时与NT-proBNP、左心室射血分数相结合,发现miR-652水平甚至有助于心肌梗死后的风险分层 (118)。在359名AMI患者与30名健康志愿者的研究中,miR-328和miR-134水平在心肌梗死后6个月内死亡或心衰的风险分层中的结果与之前研究相似 (119)。
到目前为止,唯一已知的基于人群检测miRNAs 对于心肌梗死的预测价值的前瞻性队列研究是Bruneck研究,此研究包含820例病人(27)。多变量Cox回归分析发现三个miRNA的标志物(miR-126,miR-197和miR-223)参与MI预测,miR-126水平呈正相关,miR-197和miR-223与可疑MI呈负相关,这些数据证实miRNA可用来对CAD进行基于人群的风险评估。
CAD的miRNA标志物
除了单个miRNA的研究,几个研究组研究了miRNA标志物对冠状动脉疾病的预测潜能(见表1 和 2)。
Ren等人在一组13例患有UAP和CAD与13例NCCP患者的研究中发现一簇miRNAs包括106b/25簇,miR-17 / 92A簇,miR-21 / 590-5p家族,miR-451与miR-126均发生失衡性改变(108)。重要的是,即使校正风险因素和使用他汀类药物和抗血小板药物后,这些循环miRNAs的水平仍与UAP有独立的相关性。
D’Alessandra等人发现与健康对照组比较,特定血浆的miRNA标志物能够有效地诊断稳定型心绞痛或不稳定型心绞痛。在一个与20名健康人相对照的53名患有SAP或患有UAP病患者研究中,作者发现两组具有鉴别SAP和UAP的miRNAs(107)。与对照组相比,SAP患者有较高水平的miR-1,miR-126和mir-485-3p,而UAP患者有较高水平的miR-1, miR-126 and miR-133a 。
AMI的miRNA标志物
李及其同事研究了117例AMI患者、182例AP患者和100例健康对照者的血清样本 (97)。在初步筛选的miRNA表达验证阶段后,他们发现一个由6个miRNA (miR-1, miR-134, miR-186, miR-208, miR-223 and miR-499) 组成的miRNA标志物组,诊断的AMI药-时曲线下总面积(AUC = 0.83),高于心脏肌钙蛋白T(AUC = 0.768)和肌酸激酶同工酶(0.709)。相比单个miRNA,miRNA组在诊断MI上有很大的优势。同样地, Meder 等人也证实相似的结果,与对照组相比较,AMI组病人全血中的121个miRNA发生变化(120)。作者发现与单个miRNA相比,一组由20个miRNA组成的标志物组预测AMI具有更好的特异性和敏感性以及较高的预测能力。他们的研究甚至应用了miRNA作为AMI的早期诊断指标而不是TnT。同一研究组在18个AMI患者中进行miRNA全基因的动力学研究以及对miRNA的表达水平进行连续测量(121)。他们找到了在AMI早期发生变化的一组由7个miRNA组成的miRNA标志物组(miR-636, miR-7-1*,miR-380*,miR-1254,miR-455-3p,miR-56和 miR-1291) ,其中的5个miRNA是之前发现的由20个miRNA组成的标志物组的一部分。 表1为心肌梗死中miRNA的概述。
虽然这些研究的样本量小,但是他们强调了miRNA作为AMI诊断的生物标志物的潜能。
疾病治疗的miRNA
miRNA调控功能的重要性常常在应激条件下被证实,同时miRNA功能的失调可能导致外在轻微变化(66)。在治疗上应用miRNA的困难在于单个的miRNA能够影响多种蛋白和基因的表达,而与此同时受调控的miRNA可以涉及大量不同的miRNA。单个miRNA对靶蛋白及基因的作用较微弱,而单个miRNA受大量miRNA影响,表达的细胞蛋白组弥补了单个miRNA的不足(122)。而且,miRSNPs 既能改变miRNA自身的序列也能改变miRNA靶点的序列,这不仅关系到癌症风险和预后还改变了治疗反应(24)。然而,体外实验和动物模型的预期结果表明成功应用miRNA作为治疗靶点的可能性。
涉及miRNA的治疗方案表明在某种疾病中miRNA水平的特定影响。根据病理生理学机制,其影响包括抑制miRNAs以及提高miRNA水平或将它们复制取代。复制取代可以通过miRNA模拟技术实现(mir模拟),是一种采用双链RNA人工合成miRNA类似片段的基因沉默方法(123)。这些RNA片段“模仿”内源性miRNA并且专门与靶mRNA结合,激活RNA诱导的沉默复合物(RNA-induced silencing complex,RISC),下调特定mRNAs,从而诱导基因抑制(图1)。相对于内源性miRNA,mir模拟物发挥着基因特异性的作用 (123)。在癌症方面mir模拟物已经大量被研究,然而,到目前为止,在心血管疾病中却很少有研究发表。
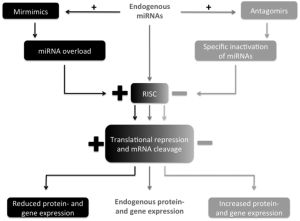
相反,miRNA的抑制可以通过antagomirs获得 (124)。antagomirs是一类化学合成的寡核苷酸特异性沉默的单链内源性miRNA(125)。Antagomirs竞争性抑制miRNA结合靶标志物(124),从而能减少RISC的激活,导致特定mRNA及基因表达的上调(图1)(126)。
进一步减少miRNA数量和影响miRNA的方法具体涉及到miRNA海绵的使用(也被称为“目标的模仿”),用来掩盖及清除。海绵基于抑制miRNA活性的机制,通过一个包含miRNA家族结合位点的竞争性抑制剂来抑制miRNA的活性 (127-129)。抑制剂可以是瞬间被质粒转染表达的RNA也可以是由稳定的染色体插入表达(127)。miRNA通过与mirRNA海绵完整或不完整的结合,mirRNA对mRNA的影响及基因表达是减弱的,因为已结合的mirRNA无法与mRNA的结合位点结合(124)。相对于antagomirs,海绵是专门只对应一个miRNA的种子区,从而能够阻止整个家族miRNA(130)。
掩盖方法是通过应用完全匹配的寡核苷酸来掩盖靶向miRNA的mRNA的结合位点,从而阻碍特定的miRNA与结合位点结合(131)。因此,少量的miRNA与靶目标相结合。清除是与特定的miRNA互补的寡核苷酸,通过与miRNA结合抑制其内源性功能(124)。
包含mirRNA模拟物的研究
研究表明miR-1的过度表达诱导凋亡,miR-1水平与大鼠心肌梗死的面积大小密切相关(55,64)。有报道表明缺血预适应增加miR-1水平,从而导致心肌梗死亡率显著减少(79)。miR-1模拟物研究表明在糖尿病中miR-1调节内皮素-1(132) ,而在心肌梗死中尚未有报道。
在诱发大鼠心肌梗死模型的梗死心肌区域中,miR-21水平减少,董和他的同事们通过缺血预适应抑制miR-21的下调,从而增加心肌梗死前miR-21的水平 (62)。同时,通过腺病毒转染表达的miR-21 减少了细胞凋亡,减少梗死面积,加快左心室重建。而且,报道称在小鼠心内注射miR-21连同miR-1和miR-24减少了心肌梗塞面积(133)。
黄和同事提出了一个不同的提高miRNA水平的治疗方法。他们采用小鼠模型与对照组相比,过度表达miR-1的间充质干细胞转染到梗死的心肌细胞后通过增强心肌细胞分化从而改善心肌功能(134)。
包含antagomir的研究
在一个心肌梗死的小鼠模型中系统应用miR-92a的抑制剂,结果抑制miR-92a的antagomir通过增强血管生长恢复了组织的损伤 (47)。miR-92a抑制剂可以诱导抗动脉粥样硬化作用以及抑制血管内皮细胞的损伤。一个体外诱导的大鼠心肌梗死模型显示mir-320的沉默减少了细胞的死亡及凋亡。应用mir-320抑制剂antagomir-320甚至显著减少了心肌梗死的梗死面积(135)。
另一研究小组在小鼠的心肌缺血再灌注模型中应用miR-29的抑制剂,从而预防了心肌细胞受损(136)。作者甚至成功地应用miR-29a与miR-29c的抑制剂减少了心肌梗死的面积。在模型中,系统的应用miR-92a抑制可以增强损伤组织的功能恢复,减少细胞调亡和改善心肌梗死面积(47)。杨等人采用诱导心肌梗死后的大鼠模型证实消除miR-1的抗抑制剂能起到抗心律失常作用(56)。
这些研究表明在CAD和MI及临床试验中使用miRNA作为治疗靶点备受关注。
分析考虑和标准化
一些混杂因素可以影响miRNA数据的质量,需要审慎考虑。目前我们仍采用非标准化的模式进行研究,不同的样品制备方法,检测及标准化策略,因而需要制定一个通用的参考标准。而且,分析因素引起的变化可以大大的影响miRNA数据(a HREF="#B137">137)。这种变化可能在临床情况下受到特别的挑战,因为不同临床中心在样品采集,样品处理和性能检测中也存在差异。因此,我们需要消除技术和分析变异以及避免人工数据的产生,需要对所有步骤的标准方法达成共识。在这里,我们简要讨论主要的影响因素。
方法学的挑战及其影响因素
RNA分离
由于体液中的miRNA浓度很低,我们将期望赋予在miRNA检测效率上。由于极少数量的抑制剂提取也会影响miRNA的提取数量。因此,找到以最大限度的重复性及可靠性的miRNA分离方法是十分关键的,一些研究已经描述不同质量的用来分离miRNA的商业试剂盒 (138).
检测方法
高通量测序,实时定量PCR(qPCR)和微阵列技术为目前主要应用的三种定量方法。由于高度特异性及敏感性,定量PCR被认定为miRNA组的检测及定量分析的金标准。高通量测序miRNA在新的miRNA的发现中具有优越性。微阵列技术被认为是一个更大范围的标准化的全基因组分析的最佳选择(139)。在过去的几年中,miRNA定量检测方法已普遍适用,科学家能够容易地应用qRT-PCR技术定量 miRNA的水平。
然而,miRNA的检测相比于以蛋白质为基础的生物标志物的检测,仍然缺乏标准化的方法和自动化工作流程以及明确的指南指出哪种体液使用哪种类型的miNA。
标准化
miRNA定量的一个重要技术方面在于标准化的和可重复性的分开方法及检测计划。同时,为了有效得出可靠、可重复的结果也需要统一的分析及标准化方法。不同标准化方法对miRNA定量检测的影响已经阐述(140)。我们未采取标准化的规范,因此产生了各种标准化策略(141)。一个被广泛应用的方法是基于C线虫miR-39穗来作为一个miRNA定量参考。另一方面,值得高度推荐的标准应适用于所有测量的miRNA平均值以及在相似水平检测下的个体miRNA (142)。
血清与细胞质中miRNA的不同水平比较
一些作者发现血浆与细胞质中的miRNA相似处(7,25),但是也发现二者中miRNA的不同之处(143)。值得注意的是,在细胞质中的血小板相关的miRNA的浓度远远高于血浆中的miRNA(73)。由于基于miRNA的CAD诊断的领域中,血小板衍生的miRNA(如miR-126, miR-223 和miR-197)在未知心血管事件的预测中似乎发挥重要的作用(27)。在未来临床miRNA定量研究领域中,标准化的方法是有必要的。
影响miRNA的参数水平
以前的研究已经证实采血前给予患者肝素会干扰miRNA的定量结果(144,145)。因此,在选择患者进行体内miRNA研究时应高度警惕血样中肝素提前给药的问题。
CAD中miRNA定量也可能因类似他汀类药物和血管紧张素转换酶抑制剂等药物的摄入而变化(102)。
这些研究结果强调了临床上对miRNA的药物和代谢物相关影响的重要性。同时,数据的不一致反映了我们需要进一步研究miRNA水平在循环血液中受什么影响。
此外,需考虑到的是,生物标志物会受到清除速率的影响。Gidlöf等人发现心脏miRNA水平与肾功能有很大关系,这表明肾功能也可能影响细胞质中miRNA的水平(84)。
未来的计划
尽管我们对miRNA在心血管疾病的诊断和预后中所扮演的角色产生浓厚的兴趣,但是我们只触及了它复杂性的表面。为了充分阐明miRNA的潜能,我们需要强大的样品处理能力、先进的科技和分析方法。
新miRNAs的发现和确认的已报道出的结果应该是大型研究网络的一个组成部分,涉及的行业和专家处在不同的领域,如分子生物学,分析化学,生物信息学,临床试验设计,流行病学,统计学以及保健经济学(146)。为了践行一些生物标志物研究的结果,几个合作性倡议在近几年已经施行了。一个是欧盟资助的BiomarCaRE联盟(欧洲生物标志物的心血管风险评估),它是欧洲的一个合作研究项目,入选来自13个欧洲国家的临床或患病人群超过300000名参与者(147)。其他的miRNA的生物标志物相对于他们的CAD和MI事件的预测价值被评估。大规模的循环miRNA定量研究尚未施行,旨在进行评价目前已经开展的单个miRNA的研究结果。
领域内的新玩家:长链非编码RNA
除了miRNA,最近长链非编码RNA(long non-coding RNAs,lncRNA)在基因调控领域的作用日益受到重视 (148)。一个由大于200个核苷酸组成的lncRNAs参与基因表达的调控机制(149)。循环中的lncRNAs被作为心血管疾病的生物标志物进行评价。第一批研究表明414例AMI患者的血细胞lncRNAs具有不同的表达水平(150),说明lncRNAs参与AMI病理生理过程以及有可能作为心血管生物标志物而被应用。
总结
许多研究报道了miRNA在心血管疾病领域是新的诊断和预后标志物。循环miRNA作为生物标志物应用于疾病的诊断与预后是已有的基于蛋白质标志物潜在的附加板块。体外实验和动物模型研究证实miRNA的病理生理途径,并研制基于miRNA来干预疾病的进展及其并发症的方法。mir模拟物和antagomirs在心血管领域的应用尚未找到相应的临床试验方法,但很有前景的研究结果反映了miRNA在未来治疗领域应用中的潜在可能性。
我们对参与miRNA和基因表达的复杂的调控机制尚未完全理解。与此同时,体液中miRNA检测和定量的技术方面(如RNA的规范化和生物材料)有必要进行基于可重复性方案的标准化。
致谢
资金:T Zeller感谢欧洲联盟(biomarcare,批准号:health-2011-278913), the Deutsche Stiftung für Herzforschung 和 the Bundesministerium für Bildung und Forschung的基金资助。
声明: C Schulte宣呈无利益冲突。
References
- Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell 1993;75:843-54. [PubMed]
- Lagos-Quintana M, Rauhut R, Lendeckel W, et al. Identification of novel genes coding for small expressed RNAs. Science 2001;294:853-8. [PubMed]
- Reid G, Kirschner MB, van Zandwijk N. Circulating microRNAs: Association with disease and potential use as biomarkers. Crit Rev Oncol Hematol 2011;80:193-208. [PubMed]
- Chandrasekaran K, Karolina DS, Sepramaniam S, et al. Role of microRNAs in kidney homeostasis and disease. Kidney Int 2012;81:617-27. [PubMed]
- Fan HM, Sun XY, Guo W, et al. Differential expression of microRNA in peripheral blood mononuclear cells as specific biomarker for major depressive disorder patients. J Psychiatr Res 2014;59:45-52. [PubMed]
- Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes Dev 2001;15:188-200. [PubMed]
- Fichtlscherer S, De Rosa S, Fox H, et al. Circulating microRNAs in patients with coronary artery disease. Circ Res 2010;107:677-84. [PubMed]
- Small EM, Frost RJ, Olson EN. MicroRNAs add a new dimension to cardiovascular disease. Circulation 2010;121:1022-32. [PubMed]
- Corsten MF, Dennert R, Jochems S, et al. Circulating MicroRNA-208b and MicroRNA-499 reflect myocardial damage in cardiovascular disease. Circ Cardiovasc Genet 2010;3:499-506. [PubMed]
- Gomes da Silva AM, Silbiger VN. miRNAs as biomarkers of atrial fibrillation. Biomarkers 2014;19:631-6. [PubMed]
- Lau NC, Lim LP, Weinstein EG, et al. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans. Science 2001;294:858-62. [PubMed]
- Reinhart BJ, Weinstein EG, Rhoades MW, et al. MicroRNAs in plants. Genes Dev 2002;16:1616-26. [PubMed]
- Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 2004;116:281-97. [PubMed]
- Ambros V, Bartel B, Bartel DP, et al. A uniform system for microRNA annotation. Rna 2003;9:277-9. [PubMed]
- Farh KK, Grimson A, Jan C, et al. The widespread impact of mammalian MicroRNAs on mRNA repression and evolution. Science 2005;310:1817-21. [PubMed]
- Kozomara A, Griffiths-Jones S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Res 2014;42:D68-73. [PubMed]
- Friedman RC, Farh KK, Burge CB, et al. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res 2009;19:92-105. [PubMed]
- Bushati N, Cohen SM. microRNA functions. Annu Rev Cell Dev Biol 2007;23:175-205. [PubMed]
- van Rooij E. The art of microRNA research. Circ Res 2011;108:219-34. [PubMed]
- Bauersachs J, Thum T. Biogenesis and regulation of cardiovascular microRNAs. Circ Res 2011;109:334-47. [PubMed]
- Creemers EE, Tijsen AJ, Pinto YM. Circulating microRNAs: novel biomarkers and extracellular communicators in cardiovascular disease? Circ Res 2012;110:483-95. [PubMed]
- Dzikiewicz-Krawczyk A. MicroRNA polymorphisms as markers of risk, prognosis and treatment response in hematological malignancies. Crit Rev Oncol Hematol 2015;93:1-17. [PubMed]
- Mishra PJ, Bertino JR. MicroRNA polymorphisms: the future of pharmacogenomics, molecular epidemiology and individualized medicine. Pharmacogenomics 2009;10:399-416. [PubMed]
- Salzman DW, Weidhaas JB. SNPing cancer in the bud: microRNA and microRNA-target site polymorphisms as diagnostic and prognostic biomarkers in cancer. Pharmacol Ther 2013;137:55-63. [PubMed]
- Mitchell PS, Parkin RK, Kroh EM, et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proc Natl Acad Sci U S A 2008;105:10513-8. [PubMed]
- Weber JA, Baxter DH, Zhang S, et al. The microRNA spectrum in 12 body fluids. Clin Chem 2010;56:1733-41. [PubMed]
- Zampetaki A, Willeit P, Tilling L, et al. Prospective study on circulating MicroRNAs and risk of myocardial infarction. J Am Coll Cardiol 2012;60:290-9. [PubMed]
- Contu R, Latronico MV, Condorelli G. Circulating microRNAs as potential biomarkers of coronary artery disease: a promise to be fulfilled? Circ Res 2010;107:573-4. [PubMed]
- Wu AH, Jaffe AS. The clinical need for high-sensitivity cardiac troponin assays for acute coronary syndromes and the role for serial testing. Am Heart J 2008;155:208-14. [PubMed]
- Keller T, Zeller T, Peetz D, et al. Sensitive troponin I assay in early diagnosis of acute myocardial infarction. N Engl J Med 2009;361:868-77. [PubMed]
- Hamm CW, Bassand JP, Agewall S, et al. ESC Guidelines for the management of acute coronary syndromes in patients presenting without persistent ST-segment elevation: The Task Force for the management of acute coronary syndromes (ACS) in patients presenting without persistent ST-segment elevation of the European Society of Cardiology (ESC). Eur Heart J 2011;32:2999-3054. [PubMed]
- Li C, Pei F, Zhu X, et al. Circulating microRNAs as novel and sensitive biomarkers of acute myocardial Infarction. Clin Biochem 2012;45:727-32. [PubMed]
- Wang R, Li N, Zhang Y, et al. Circulating microRNAs are promising novel biomarkers of acute myocardial infarction. Intern Med 2011;50:1789-95. [PubMed]
- Kuwabara Y, Ono K, Horie T, et al. Increased microRNA-1 and microRNA-133a levels in serum of patients with cardiovascular disease indicate myocardial damage. Circ Cardiovasc Genet 2011;4:446-54. [PubMed]
- Jaguszewski M, Osipova J, Ghadri JR, et al. A signature of circulating microRNAs differentiates takotsubo cardiomyopathy from acute myocardial infarction. Eur Heart J 2014;35:999-1006. [PubMed]
- Suárez Y, Fernández-Hernando C, Pober JS, et al. Dicer dependent microRNAs regulate gene expression and functions in human endothelial cells. Circ Res 2007;100:1164-73. [PubMed]
- Kuehbacher A, Urbich C, Zeiher AM, et al. Role of Dicer and Drosha for endothelial microRNA expression and angiogenesis. Circ Res 2007;101:59-68. [PubMed]
- Schroen B, Heymans S. Small but smart--microRNAs in the centre of inflammatory processes during cardiovascular diseases, the metabolic syndrome, and ageing. Cardiovasc Res 2012;93:605-13. [PubMed]
- Jovanović I, Zivković M, Jovanović J, et al. The co-inertia approach in identification of specific microRNA in early and advanced atherosclerosis plaque. Med Hypotheses 2014;83:11-5. [PubMed]
- Menghini R, Stohr R, Federici M. MicroRNAs in vascular aging and atherosclerosis. Ageing Res Rev 2014;17:68-78. [PubMed]
- Urbich C, Kuehbacher A, Dimmeler S. Role of microRNAs in vascular diseases, inflammation, and angiogenesis. Cardiovasc Res 2008;79:581-8. [PubMed]
- Chen Y, Gorski DH. Regulation of angiogenesis through a microRNA (miR-130a) that down-regulates antiangiogenic homeobox genes GAX and HOXA5. Blood 2008;111:1217-26. [PubMed]
- Tuccoli A, Poliseno L, Rainaldi G. miRNAs regulate miRNAs: coordinated transcriptional and post-transcriptional regulation. Cell Cycle 2006;5:2473-6. [PubMed]
- Linton MF, Fazio S. Macrophages, inflammation, and atherosclerosis. Int J Obes Relat Metab Disord 2003;27 Suppl 3:S35-40. [PubMed]
- Vickers KC, Landstreet SR, Levin MG, et al. MicroRNA-223 coordinates cholesterol homeostasis. Proc Natl Acad Sci U S A 2014;111:14518-23. [PubMed]
- Zhang J, Ren J, Chen H, et al. Inflammation induced-endothelial cells release angiogenesis associated-microRNAs into circulation by microparticles. Chin Med J (Engl) 2014;127:2212-7. [PubMed]
- Bonauer A, Carmona G, Iwasaki M, et al. MicroRNA-92a controls angiogenesis and functional recovery of ischemic tissues in mice. Science 2009;324:1710-3. [PubMed]
- Wu W, Xiao H, Laguna-Fernandez A, et al. Flow-Dependent Regulation of Kruppel-Like Factor 2 Is Mediated by MicroRNA-92a. Circulation 2011;124:633-41. [PubMed]
- Dahan D, Ekman M, Larsson-Callerfelt AK, et al. Induction of angiotensin converting enzyme after miR-143/145 deletion is critical for impaired smooth muscle contractility. Am J Physiol Cell Physiol 2014;307:C1093-101. [PubMed]
- Elia L, Quintavalle M, Zhang J, et al. The knockout of miR-143 and -145 alters smooth muscle cell maintenance and vascular homeostasis in mice: correlates with human disease. Cell Death Differ 2009;16:1590-8. [PubMed]
- Xin M, Small EM, Sutherland LB, et al. MicroRNAs miR-143 and miR-145 modulate cytoskeletal dynamics and responsiveness of smooth muscle cells to injury. Genes Dev 2009;23:2166-78. [PubMed]
- Kumar S, Kim CW, Simmons RD, et al. Role of flow-sensitive microRNAs in endothelial dysfunction and atherosclerosis: mechanosensitive athero-miRs. Arterioscler Thromb Vasc Biol 2014;34:2206-16. [PubMed]
- Zhao Y, Samal E, Srivastava D. Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis. Nature 2005;436:214-20. [PubMed]
- Ikeda S, He A, Kong SW, et al. MicroRNA-1 negatively regulates expression of the hypertrophy-associated calmodulin and Mef2a genes. Mol Cell Biol 2009;29:2193-204. [PubMed]
- Xu C, Lu Y, Pan Z, et al. The muscle-specific microRNAs miR-1 and miR-133 produce opposing effects on apoptosis by targeting HSP60, HSP70 and caspase-9 in cardiomyocytes. J Cell Sci 2007;120:3045-52. [PubMed]
- Yang B, Lin H, Xiao J, et al. The muscle-specific microRNA miR-1 regulates cardiac arrhythmogenic potential by targeting GJA1 and KCNJ2. Nat Med 2007;13:486-91. [PubMed]
- Bostjancic E, Zidar N, Stajner D, et al. MicroRNA miR-1 is up-regulated in remote myocardium in patients with myocardial infarction. Folia Biol (Praha) 2010;56:27-31. [PubMed]
- Chen JF, Mandel EM, Thomson JM, et al. The role of microRNA-1 and microRNA-133 in skeletal muscle proliferation and differentiation. Nat Genet 2006;38:228-33. [PubMed]
- Ye Y, Perez-Polo JR, Qian J, et al. The role of microRNA in modulating myocardial ischemia-reperfusion injury. Physiol Genomics 2011;43:534-42. [PubMed]
- van Rooij E, Sutherland LB, Qi X, et al. Control of stress-dependent cardiac growth and gene expression by a microRNA. Science 2007;316:575-9. [PubMed]
- van Rooij E, Sutherland LB, Thatcher JE, et al. Dysregulation of microRNAs after myocardial infarction reveals a role of miR-29 in cardiac fibrosis. Proc Natl Acad Sci U S A 2008;105:13027-32. [PubMed]
- Dong S, Cheng Y, Yang J, et al. MicroRNA expression signature and the role of microRNA-21 in the early phase of acute myocardial infarction. J Biol Chem 2009;284:29514-25. [PubMed]
- Wang S, Aurora AB, Johnson BA, et al. The endothelial-specific microRNA miR-126 governs vascular integrity and angiogenesis. Dev Cell 2008;15:261-71. [PubMed]
- Tang Y, Zheng J, Sun Y, et al. MicroRNA-1 regulates cardiomyocyte apoptosis by targeting Bcl-2. Int Heart J 2009;50:377-87. [PubMed]
- Bostjancic E, Zidar N, Stajer D, et al. MicroRNAs miR-1, miR-133a, miR-133b and miR-208 are dysregulated in human myocardial infarction. Cardiology 2010;115:163-9. [PubMed]
- Mendell JT, Olson EN. MicroRNAs in stress signaling and human disease. Cell 2012;148:1172-87. [PubMed]
- Boon RA, Vickers KC. Intercellular transport of microRNAs. Arterioscler Thromb Vasc Biol 2013;33:186-92. [PubMed]
- Simons M, Raposo G. Exosomes--vesicular carriers for intercellular communication. Curr Opin Cell Biol 2009;21:575-81. [PubMed]
- Zernecke A, Bidzhekov K, Noels H, et al. Delivery of microRNA-126 by apoptotic bodies induces CXCL12-dependent vascular protection. Sci Signal 2009;2:ra81. [PubMed]
- Valadi H, Ekström K, Bossios A, et al. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat Cell Biol 2007;9:654-9. [PubMed]
- Arroyo JD, Chevillet JR, Kroh EM, et al. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc Natl Acad Sci U S A 2011;108:5003-8. [PubMed]
- Vickers KC, Palmisano BT, Shoucri BM, et al. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nat Cell Biol 2011;13:423-33. [PubMed]
- Chen X, Ba Y, Ma L, et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res 2008;18:997-1006. [PubMed]
- Ji X, Takahashi R, Hiura Y, et al. Plasma miR-208 as a biomarker of myocardial injury. Clin Chem 2009;55:1944-9. [PubMed]
- Wang GK, Zhu JQ, Zhang JT, et al. Circulating microRNA: a novel potential biomarker for early diagnosis of acute myocardial infarction in humans. Eur Heart J 2010;31:659-66. [PubMed]
- Gidlöf O, Smith JG, Miyazu K, et al. Circulating cardio-enriched microRNAs are associated with long-term prognosis following myocardial infarction. BMC Cardiovasc Disord 2013;13:12. [PubMed]
- Adachi T, Nakanishi M, Otsuka Y, et al. Plasma microRNA 499 as a biomarker of acute myocardial infarction. Clin Chem 2010;56:1183-5. [PubMed]
- Olivieri F, Antonicelli R, Lorenzi M, et al. Diagnostic potential of circulating miR-499-5p in elderly patients with acute non ST-elevation myocardial infarction. Int J Cardiol 2013;167:531-6. [PubMed]
- Cheng Y, Tan N, Yang J, et al. A translational study of circulating cell-free microRNA-1 in acute myocardial infarction. Clin Sci (Lond) 2010;119:87-95. [PubMed]
- Ai J, Zhang R, Li Y, et al. Circulating microRNA-1 as a potential novel biomarker for acute myocardial infarction. Biochem Biophys Res Commun 2010;391:73-7. [PubMed]
- Widera C, Gupta SK, Lorenzen JM, et al. Diagnostic and prognostic impact of six circulating microRNAs in acute coronary syndrome. J Mol Cell Cardiol 2011;51:872-5. [PubMed]
- Li YQ, Zhang MF, Wen HY, et al. Comparing the diagnostic values of circulating microRNAs and cardiac troponin T in patients with acute myocardial infarction. Clinics (Sao Paulo) 2013;68:75-80. [PubMed]
- D’Alessandra Y, Devanna P, Limana F, et al. Circulating microRNAs are new and sensitive biomarkers of myocardial infarction. Eur Heart J 2010;31:2765-73. [PubMed]
- Gidlöf O, Andersson P, van der Pals J, et al. Cardiospecific microRNA plasma levels correlate with troponin and cardiac function in patients with ST elevation myocardial infarction, are selectively dependent on renal elimination, and can be detected in urine samples. Cardiology 2011;118:217-26. [PubMed]
- Duan XX, Zhou X, Wang XB, et al. Urine cardiac specific microRNA-1 level in patients with ST segment elevation acute myocardial infarction. Zhongguo Wei Zhong Bing Ji Jiu Yi Xue 2012;24:709-12. [PubMed]
- Long G, Wang F, Duan Q, et al. Human circulating microRNA-1 and microRNA-126 as potential novel indicators for acute myocardial infarction. Int J Biol Sci 2012;8:811-8. [PubMed]
- Li LM, Cai WB, Ye Q, et al. Comparison of plasma microRNA-1 and cardiac troponin T in early diagnosis of patients with acute myocardial infarction. World J Emerg Med 2014;5:182-6. [PubMed]
- Hsu A, Chen SJ, Chang YS, et al. Systemic approach to identify serum microRNAs as potential biomarkers for acute myocardial infarction. Biomed Res Int 2014;2014:418628.
- Long G, Wang F, Duan Q, et al. Circulating miR-30a, miR-195 and let-7b associated with acute myocardial infarction. PLoS One 2012;7:e50926. [PubMed]
- De Rosa S, Fichtlscherer S, Lehmann R, et al. Transcoronary concentration gradients of circulating microRNAs. Circulation 2011;124:1936-44. [PubMed]
- Wang F, Long G, Zhao C, et al. Plasma microRNA-133a is a new marker for both acute myocardial infarction and underlying coronary artery stenosis. J Transl Med 2013;11:222. [PubMed]
- Peng L, Chun-guang Q, Bei-fang L, et al. Clinical impact of circulating miR-133, miR-1291 and miR-663b in plasma of patients with acute myocardial infarction. Diagn Pathol 2014;9:89. [PubMed]
- Devaux Y, Vausort M, McCann GP, et al. MicroRNA-150: a novel marker of left ventricular remodeling after acute myocardial infarction. Circ Cardiovasc Genet 2013;6:290-8. [PubMed]
- Devaux Y, Vausort M, Goretti E, et al. Use of circulating microRNAs to diagnose acute myocardial infarction. Clin Chem 2012;58:559-67. [PubMed]
- Nabiałek E, Wańha W, Kula D, et al. Circulating microRNAs (miR-423-5p, miR-208a and miR-1) in acute myocardial infarction and stable coronary heart disease. Minerva Cardioangiol 2013;61:627-37. [PubMed]
- Olivieri F, Antonicelli R, Spazzafumo L, et al. Admission levels of circulating miR-499-5p and risk of death in elderly patients after acute non-ST elevation myocardial infarction. Int J Cardiol 2014;172:e276-8. [PubMed]
- Li C, Fang Z, Jiang T, et al. Serum microRNAs profile from genome-wide serves as a fingerprint for diagnosis of acute myocardial infarction and angina pectoris. BMC Med Genomics 2013;6:16. [PubMed]
- Devaux Y, Vausort M, McCann GP, et al. A panel of 4 microRNAs facilitates the prediction of left ventricular contractility after acute myocardial infarction. PLoS One 2013;8:e70644. [PubMed]
- Cheng C, Wang Q, You W, et al. MiRNAs as biomarkers of myocardial infarction: a meta-analysis. PLoS One 2014;9:e88566. [PubMed]
- Hoekstra M, van der Lans CA, Halvorsen B, et al. The peripheral blood mononuclear cell microRNA signature of coronary artery disease. Biochem Biophys Res Commun 2010;394:792-7. [PubMed]
- Taurino C, Miller WH, McBride MW, et al. Gene expression profiling in whole blood of patients with coronary artery disease. Clin Sci (Lond) 2010;119:335-43. [PubMed]
- Weber M, Baker MB, Patel RS, et al. MicroRNA Expression Profile in CAD Patients and the Impact of ACEI/ARB. Cardiol Res Pract 2011;2011:532915.
- Zhu J, Chen T, Yang L, et al. Regulation of microRNA-155 in atherosclerotic inflammatory responses by targeting MAP3K10. PLoS One 2012;7:e46551. [PubMed]
- Xiong XD, Cho M, Cai XP, et al. A common variant in pre-miR-146 is associated with coronary artery disease risk and its mature miRNA expression. Mutat Res Fundam Mol Mech Mutagen 2014;761:15-20. [PubMed]
- Ramkaran P, Khan S, Phulukdaree A, et al. miR-146a polymorphism influences levels of miR-146a, IRAK-1, and TRAF-6 in young patients with coronary artery disease. Cell Biochem Biophys 2014;68:259-66. [PubMed]
- Sondermeijer BM, Bakker A, Halliani A, et al. Platelets in patients with premature coronary artery disease exhibit upregulation of miRNA340* and miRNA624*. PLoS One 2011;6:e25946. [PubMed]
- D’Alessandra Y, Carena MC, Spazzafumo L, et al. Diagnostic potential of plasmatic MicroRNA signatures in stable and unstable angina. PLoS One 2013;8:e80345. [PubMed]
- Ren J, Zhang J, Xu N, et al. Signature of circulating microRNAs as potential biomarkers in vulnerable coronary artery disease. PLoS One 2013;8:e80738. [PubMed]
- Zhang Q, Kandic I, Kutryk MJ. Dysregulation of angiogenesis-related microRNAs in endothelial progenitor cells from patients with coronary artery disease. Biochem Biophys Res Commun 2011;405:42-6. [PubMed]
- Jansen F, Yang X, Proebsting S, et al. MicroRNA Expression in Circulating Microvesicles Predicts Cardiovascular Events in Patients With Coronary Artery Disease. J Am Heart Assoc 2014;3:e001249. [PubMed]
- Takahashi Y, Satoh M, Minami Y, et al. Expression of miR-146a/b is associated with the Toll-like receptor 4 signal in coronary artery disease: effect of renin-angiotensin system blockade and statins on miRNA-146a/b and Toll-like receptor 4 levels. Clin Sci (Lond) 2010;119:395-405. [PubMed]
- Zhu GF, Yang LX, Guo RW, et al. microRNA-155 is inversely associated with severity of coronary stenotic lesions calculated by the Gensini score. Coron Artery Dis 2014;25:304-10. [PubMed]
- Lu HQ, Liang C, He ZQ, et al. Circulating miR-214 is associated with the severity of coronary artery disease. J Geriatr Cardiol 2013;10:34-8. [PubMed]
- Minami Y, Satoh M, Maesawa C, et al. Effect of atorvastatin on microRNA 221 / 222 expression in endothelial progenitor cells obtained from patients with coronary artery disease. Eur J Clin Invest 2009;39:359-67. [PubMed]
- Zeller T, Keller T, Ojeda F, et al. Assessment of microRNAs in patients with unstable angina pectoris. Eur Heart J 2014;35:2106-14. [PubMed]
- Diehl P, Fricke A, Sander L, et al. Microparticles: major transport vehicles for distinct microRNAs in circulation. Cardiovasc Res 2012;93:633-44. [PubMed]
- Xu CF, Yu CH, Li YM. Regulation of hepatic microRNA expression in response to ischemic preconditioning following ischemia/reperfusion injury in mice. Omics 2009;13:513-20. [PubMed]
- Pilbrow AP, Cordeddu L, Cameron VA, et al. Circulating miR-323-3p and miR-652: candidate markers for the presence and progression of acute coronary syndromes. Int J Cardiol 2014;176:375-85. [PubMed]
- He F, Lv P, Zhao X, et al. Predictive value of circulating miR-328 and miR-134 for acute myocardial infarction. Mol Cell Biochem 2014;394:137-44. [PubMed]
- Meder B, Keller A, Vogel B, et al. MicroRNA signatures in total peripheral blood as novel biomarkers for acute myocardial infarction. Basic Res Cardiol 2011;106:13-23. [PubMed]
- Vogel B, Keller A, Frese KS, et al. Refining diagnostic microRNA signatures by whole-miRNome kinetic analysis in acute myocardial infarction. Clin Chem 2013;59:410-8. [PubMed]
- Olson EN. MicroRNAs as therapeutic targets and biomarkers of cardiovascular disease. Sci Transl Med 2014;6:239ps3.
- Wang Z. The guideline of the design and validation of MiRNA mimics. Methods Mol Biol 2011;676:211-23. [PubMed]
- Caroli A, Cardillo MT, Galea R, et al. Potential therapeutic role of microRNAs in ischemic heart disease. J Cardiol 2013;61:315-20. [PubMed]
- Krützfeldt J, Rajewsky N, Braich R, et al. Silencing of microRNAs in vivo with ‘antagomirs’. Nature 2005;438:685-9. [PubMed]
- Oliveira-Carvalho V, Carvalho VO, Silva MM, et al. MicroRNAs: a new paradigm in the treatment and diagnosis of heart failure? Arq Bras Cardiol 2012;98:362-9. [PubMed]
- Ebert MS, Neilson JR, Sharp PA. MicroRNA sponges: competitive inhibitors of small RNAs in mammalian cells. Nat Methods 2007;4:721-6. [PubMed]
- Franco-Zorrilla JM, Valli A, Todesco M, et al. Target mimicry provides a new mechanism for regulation of microRNA activity. Nat Genet 2007;39:1033-7. [PubMed]
- Ebert MS, Sharp PA. Emerging roles for natural microRNA sponges. Curr Biol 2010;20:R858-61. [PubMed]
- Ebert MS, Sharp PA. MicroRNA sponges: progress and possibilities. Rna 2010;16:2043-50. [PubMed]
- Xiao J, Yang B, Lin H, et al. Novel approaches for gene-specific interference via manipulating actions of microRNAs: examination on the pacemaker channel genes HCN2 and HCN4. J Cell Physiol 2007;212:285-92. [PubMed]
- Feng B, Cao Y, Chen S, et al. Reprint of: miRNA-1 regulates endothelin-1 in diabetes. Life Sci 2014;118:275-80. [PubMed]
- Yin C, Salloum FN, Kukreja RC. A novel role of microRNA in late preconditioning: upregulation of endothelial nitric oxide synthase and heat shock protein 70. Circ Res 2009;104:572-5. [PubMed]
- Huang F, Li ML, Fang ZF, et al. Overexpression of MicroRNA-1 improves the efficacy of mesenchymal stem cell transplantation after myocardial infarction. Cardiology 2013;125:18-30. [PubMed]
- Ren XP, Wu J, Wang X, et al. MicroRNA-320 is involved in the regulation of cardiac ischemia/reperfusion injury by targeting heat-shock protein 20. Circulation 2009;119:2357-66. [PubMed]
- Ye Y, Hu Z, Lin Y, et al. Downregulation of microRNA-29 by antisense inhibitors and a PPAR-gamma agonist protects against myocardial ischaemia-reperfusion injury. Cardiovasc Res 2010;87:535-44. [PubMed]
- Becker N, Lockwood CM. Pre-analytical variables in miRNA analysis. Clin Biochem 2013;46:861-8. [PubMed]
- Monleau M, Bonnel S, Gostan T, et al. Comparison of different extraction techniques to profile microRNAs from human sera and peripheral blood mononuclear cells. BMC Genomics 2014;15:395. [PubMed]
- Git A, Dvinge H, Salmon-Divon M, et al. Systematic comparison of microarray profiling, real-time PCR, and next-generation sequencing technologies for measuring differential microRNA expression. Rna 2010;16:991-1006. [PubMed]
- Meyer SU, Kaiser S, Wagner C, et al. Profound effect of profiling platform and normalization strategy on detection of differentially expressed microRNAs--a comparative study. PLoS One 2012;7:e38946. [PubMed]
- Hardikar AA, Farr RJ, Joglekar MV. Circulating microRNAs: understanding the limits for quantitative measurement by real-time PCR. J Am Heart Assoc 2014;3:e000792. [PubMed]
- Zampetaki A, Mayr M. Analytical challenges and technical limitations in assessing circulating miRNAs. Thromb Haemost 2012;108:592-8. [PubMed]
- Huang X, Liang M, Dittmar R, et al. Extracellular microRNAs in urologic malignancies: chances and challenges. Int J Mol Sci 2013;14:14785-99. [PubMed]
- Kaudewitz D, Lee R, Willeit P, et al. Impact of intravenous heparin on quantification of circulating microRNAs in patients with coronary artery disease. Thromb Haemost 2013;110:609-15. [PubMed]
- Boeckel JN, Thomé CE, Leistner D, et al. Heparin selectively affects the quantification of microRNAs in human blood samples. Clin Chem 2013;59:1125-7. [PubMed]
- Siemelink MA, Zeller T. Biomarkers of coronary artery disease: the promise of the transcriptome. Curr Cardiol Rep 2014;16:513. [PubMed]
- Zeller T, Hughes M, Tuovinen T, et al. BiomarCaRE: rationale and design of the European BiomarCaRE project including 300,000 participants from 13 European countries. Eur J Epidemiol 2014;29:777-90. [PubMed]
- Skroblin P, Mayr M. “Going Long”: Long Non-Coding RNAs as Biomarkers. Circ Res 2014;115:607-9. [PubMed]
- Mercer TR, Mattick JS. Structure and function of long noncoding RNAs in epigenetic regulation. Nat Struct Mol Biol 2013;20:300-7. [PubMed]
- Vausort M, Wagner DR, Devaux Y. Long Noncoding RNAs in Patients With Acute Myocardial Infarction. Circ Res 2014;115:668-77. [PubMed]
特别感谢译者:赫嘉慧,王莹,刘越* 哈尔滨医科大学附属第一医院心内科 哈尔滨


